基于U-Net语义分割神经网络对CT医学影像进行分割
Unet:U-Net: Convolutional Networks for Biomedical Image Segmentation语义分割模型在Pytorch当中的实现
性能情况
U-Net并不适合VOC此类数据集,其更适合特征少,需要浅层特征的医药数据集(CT-soil-rock-mixture)。
| 训练数据集 | 测试数据集 | 输入图片大小 | mIOU |
|---|---|---|---|
| VOC12+SBD | VOC-Val12 | 512x512 | 55.11 |
| CT-soil-rock-mixture | CT-soil-rock-mixture | 512x512 | 92.01 |
所需环境
- scipy==1.2.1
- numpy==1.17.0
- matplotlib==3.1.2
- opencv_python==4.1.2.30
- torch==1.2.0
- torchvision==0.4.0
- tqdm==4.60.0
- Pillow==8.2.0
- h5py==2.10.0
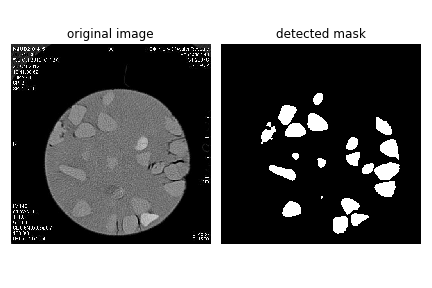
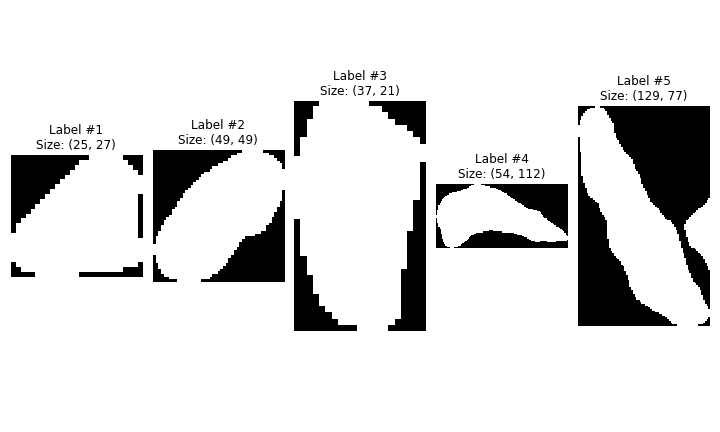
目前效果展示
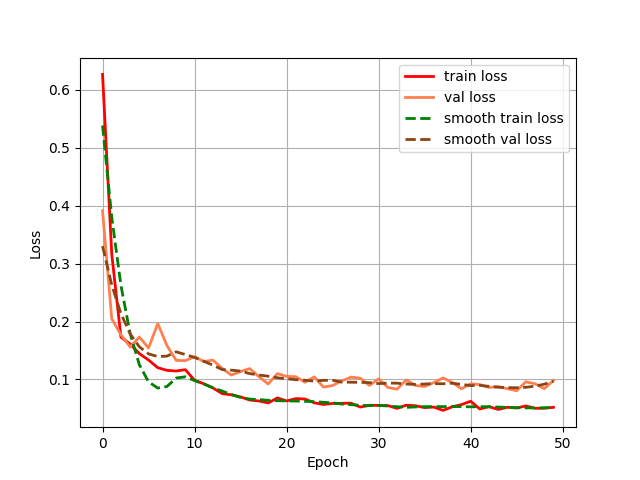
训练过程损失变化曲线图:
训练数据集说明:
格式:VOC
由于数据集标注的困难,并且无法使用之前标注了部分的Mask R-CNN 实例分割数据集,本次仅使用30张语义分割图像,数据增强到100张进行训练,得到的效果较好。
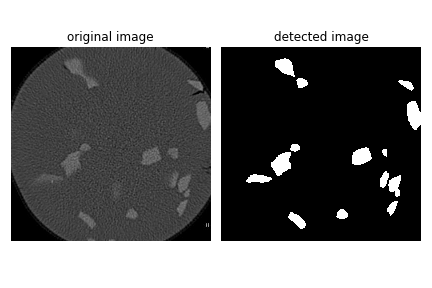
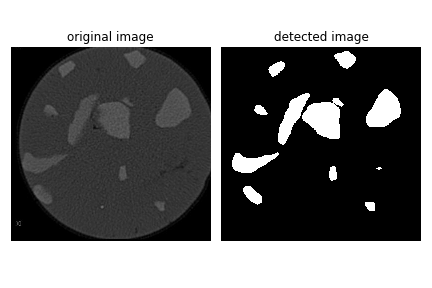
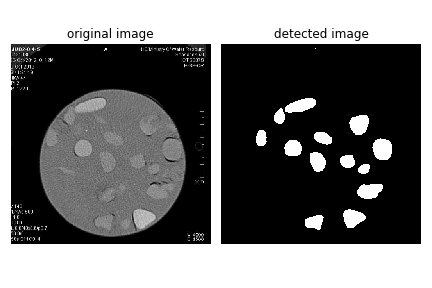
U-Net
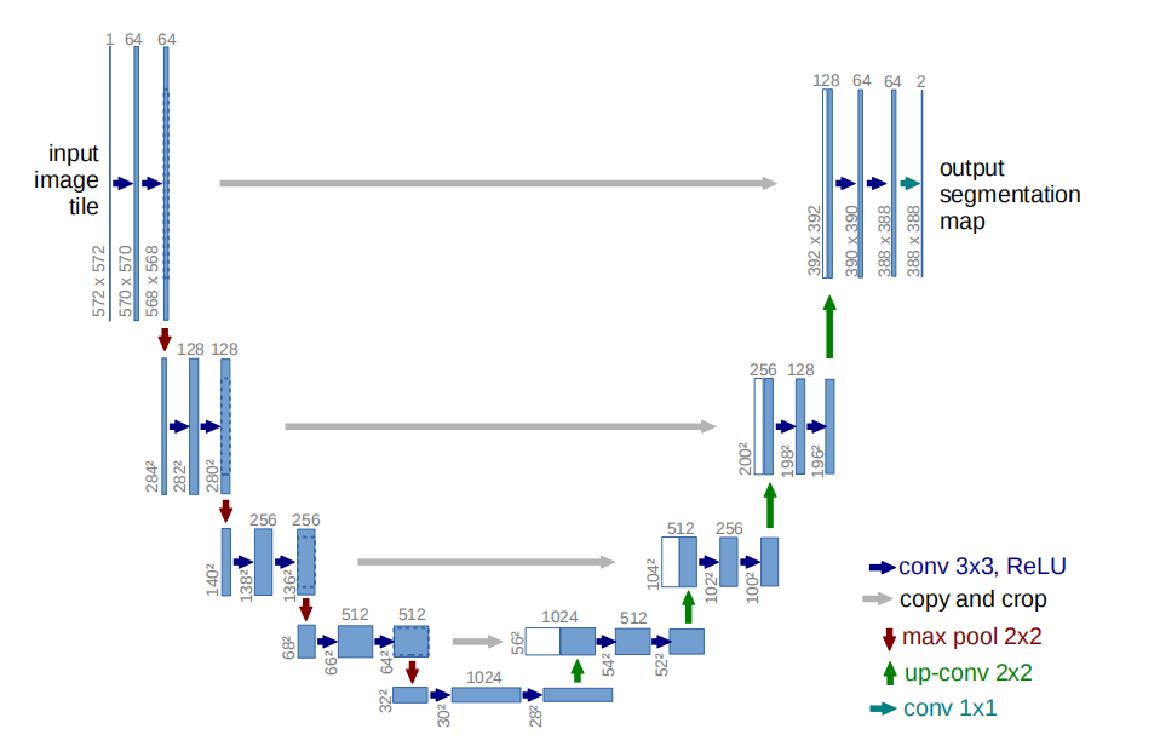
自2015年以来,在生物医学图像分割领域,U-Net得到了广泛的应用,目前已达到四千多次引用。
这个模型非常优雅,结构对称,全是卷积层,没有全连接。
因为这个网络使用更少的数据,取得更好的效果
所以我们修改并扩展这个网络。
全卷积网络其实就是一个不断缩小特征图的卷积神经网络,只不过后面的一些层中,将pooling层换为unsampling操作了。
为了使得分割结果更加精确,将高分辨率的特征图与与上采样后的低分辨率特征图进行合并。因此,分割结果就更加准确。
CT数据集特点
- 图像语义较为简单、结构较为固定。由于砾石结构固定和语义信息没有特别丰富,所以高级语义信息和低级特征都显得很重要。
- 数据量少。医学影像的数据获取相对难一些。所以我们设计的模型不宜多大,参数过多,很容易导致过拟合。
如何解决医学图像的数据非常少的问题?
- 所以需要使用大量的数据增强,意思是在不实质性的增加数据的情况下,让有限的数据产生等价于更多数据的价值。
- 数据增强:几何变换、噪声、模糊、填充、亮度等等。
如何解决目标粘连问题?
- 图像形态学操作?开运算,凸性分析……
- 使用一个加权loss,使细胞之间的缝隙有较大的训练权重,是否可行?
如何训练数据集
- 本文使用
VOC格式进行训练。 - 使用
lableme制作自己的数据集,将图片和json文件放在dataset/before中,运行json_to_dataset.py。 - 训练前将标签文件放在
VOCdevkit文件夹下的VOC2007文件夹下的SegmentationClass中。 - 训练前将图片文件放在
VOCdevkit文件夹下的VOC2007文件夹下的JPEGImages中。 - 在训练前利用
voc2unet.py文件生成对应的txt。 - 注意修改
train.py的num_classes为分类个数+1。 - 运行
train.py即可开始训练。
Reference
https://github.com/bubbliiiing/unet-pytorch
https://zhuanlan.zhihu.com/p/370931792
https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来自 Jlex の Blog!




